2023年10月,必赢线路检测3003no1•必赢线路检测3003no1公共卫生学系宋博文课题组在国际知名学术期刊Nucleic Acids Research(中科院生物一区,2022 IF=14.9)在线发表了题为“m7GHub V2.0: an updated database for decoding the N7-methylguanosine (m7G) epitranscriptome”的研究论文。该工作全面收集并解析了基于高通量测序以及第三代纳米孔测序技术得到的N7-methylguanosine(m7G)甲基化位点的定位、调控及潜在的发病机制,并构建深度学习模型探究单核苷酸多态性位点(SNP)与m7G甲基化写入(write)/抹除(erase)的动态关联。

随着N7-methylguanosine(m7G)甲基化位点测序技术的不断进展,在不同物种中发现了数万个经过实验验证的m7G位点,揭示了m7G修饰在调节包括疾病发病机制在内的众多生物过程中扮演的重要作用。 一个能够共享、注释和个性化分析m7G数据的集成资源平台将极大的促进各种生理背景下的m7G 修饰研究。在该工作中,作者构建了迄今为止针对m7G甲基化最全面的数据分析平台,用于全面收集跨多个物种的各种类型RNA中的m7G修饰,包括:(A)一个m7GDB 数据库,包含 23 个物种中鉴定的 430,898 个m7G位点,这些位点收集于广泛应用的高通量测序(NGS)和新兴的牛津纳米孔直接RNA测序(ONT)技术;(B) m7GDiseaseDB包含156,206个m7G关联突变(涉及m7G位点的写入或抹除),其中包括3,238个可能通过表观转录组干扰发挥作用的疾病相关m7G-SNPs;(C)以及两个基于深度学习模型的分析模块,用于对m7G位点 (m7GFinder) 和功能突变 (m7GSNPer) 的集合进行交互式分析。m7GHub可通过以下网址免费访问:www.rnamd.org/m7GHub2。
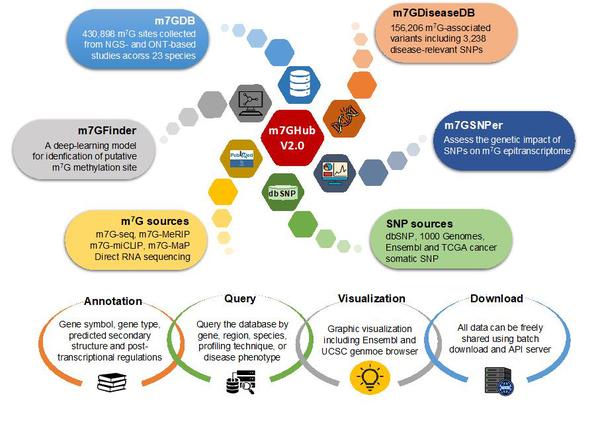
Figure 1. The overall construction of m7GHub v.2.0
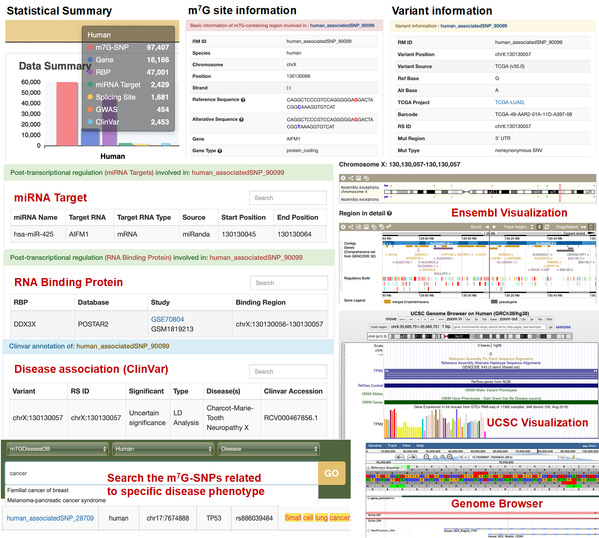
Figure 2. Enhanced web interface of m7GDiseaseDB
我校为该论文的第一完成单位,宋博文为该论文的独立通讯作者。本研究获得了国家自然科学基金;我校校特聘教授科研经费(013038030001);西交利物浦大学重点项目经费以及西交利物浦大学超算平台的资助和支持。
原文链接:https://doi.org/10.1093/nar/gkad789
(撰稿人:宋博文;审核人:关晓伟)
